Нормализиране на ДНК микрочипове
Материал от MachineLearning.
Нормализирането е важна стъпка в предварителната обработка на ДНК микрочипове, което прави възможно няколко чипа, разглеждани в експеримента, да бъдат подходящи за сравнение един с друг. Основната цел на анализа на този етап е да се изключи влиянието на системни небиологични разлики между микрочипове. Източниците на такива разлики са много: вариации в ефективността на обратната транскрипция, маркиране с багрила, хибридизация, физически разлики между чиповете, малки разлики в концентрациите на реагентите, вариации в лабораторните условия.
Показано е, че изборът на метода за нормализиране оказва значително влияние върху резултата от анализа [1] .
Съдържание
Нормализационни парадигми по отношение на избора на референтно множество
Всички методи за нормализиране под една или друга форма използват набор от гени, чиято експресия не трябва да се променя при експериментални условия, наречен референтен набор. Идеята е да се използват трансформации на данни, за да се направи разпределението на интензитета (средно, дисперсия) върху този набор еднакво за всички ДНК микрочипове.
Един подход е да се използват данни за интензивност за проби, съответстващи на така наречените домакински гени като такъв набор. По правило това са гените, необходими за поддържане на основните процеси на жизнената дейност на клетката и нивото на тяхната експресия остава приблизително постоянно при всякакви условия. Гените, предложени от Affymetrix като домакински гени, често са били използвани като референтен набор за нормализиране на ДНК микрочипове, но по-късно е установено, че при определени експериментални условия, например при изследване на определени патологии, експресията на тези гениможе да се промени значително [1] .
Повечето от използваните методи използват набора от всички гени като референтен набор, базиран на следните предположения: (1) експресията на повечето гени върху различни ДНК микрочипове е непроменена; (2) разпределението на промените в израза е близко до симетрично. Методите в този клас обикновено не са имунизирани срещу нарушения на тези допускания; в същото време те рядко се тестват на практика. Освен това не е ясно. каква част от гените може да се счита за "мнозинство" - от биологична гледна точка този въпрос не се разглежда, но от статистическа гледна точка е необходимо да се изискват поне 90% непроменени гени [1] .
Съществува и клас методи, които изграждат опорния набор директно от данните от конкретен експеримент [1] [1] . Методите от този клас зависят от наличните данни и не използват предположения (1) и (2) или позволяват директно да се посочи съотношението на гените, чиято експресия се предполага, че е непроменена от предположение (1).
Повечето от методите за нормализиране, описани по-долу, са приложими за различни начини за избор на референтен набор.
Методи за нормализиране
Мащабиране
Един от ДНК микрочиповете се избира като основен, след което всички останали се мащабират, така че техният среден интензитет да е равен на средния интензитет на основния (този метод е еквивалентен на изграждане на линейна регресия на всеки чип към базовия и след това нормализирането му с помощта на регресионна функция).
За по-голяма стабилност може да се използва подрязана средна стойност. По този начин, в стандартния софтуер на производителя на микрочипове Affymetrix, 2% от най-високите и най-ниските стойности на интензитета се отхвърлят, преди да се изчисли средната стойност. Друга модификация - мащабирането до средна интензивност не е приключилоосновен чип, но за всяка подгрупа от неговите проби, съответстваща на един ген.
Affymetrix предлага да се използва мащабиране в последната стъпка на предварителната обработка, като се прилага директно към матрици на изрази, но може да се приложи и към матрици на интензитет.
Схема за изпълнение на мащабиране
Нелинейни методи
Предложени са голям брой нелинейни методи за нормализиране на данни, като се използват различни потребителски функции, които заместват линейната регресия от предишния пример. Такива функции включват кръстосани валове >[1] , течащи средни линии [1] , изглаждане на льос [1] и др. Обикновено нелинейната нормализация се извършва върху набор от рангово-инвариантни сонди, т.е. сонди, които имат еднакъв ранг във всички микрочипове.
Блок-схема на нелинейна нормализация
Квантилна нормализация
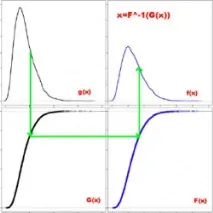
Целта на квантилната нормализация е да направи емпиричните разпределения на интензитета на всички микрочипове еднакви. За това се използва трансформация на формата, където е емпиричното разпределение на интензитетите на всеки чип, е емпиричното разпределение на интензитетите на осреднения чип. Можете да промените метода чрез оценяване и по-плавни функции. Въпреки това, за данни с голяма размерност на практика е достатъчна груба оценка.
Даденият метод за нормализация на квантилите не е единственият метод за нормализация, базиран на квантили. Описан е метод, базиран на конструиране на сплайнове чрез набор от квантили [1] , непараметричен метод за присвояване на едно и също разпределение на всеки микрочип [1] и други.
Схема за извършване на квантилна нормализация
Циклична нормализация с помощта на локална регресия (цикличен льос)
Този метод се основава на многократно прилагане на локална регресия(на английски LOWESS, LOESS) към двойки ДНК микрочипове. Процедурата изброява всички двойки комбинации от микрочипове, повтаряйки процеса на нормализиране, докато се достигне конвергенция. Един от неговите недостатъци е необходимостта от локални регресионни приложения, въпреки че обикновено са необходими само една или две стъпки на цикъл. Можете значително да намалите времето за изчисление, като използвате подмножество от данни за изграждане на регресионна зависимост.
По-долу е алгоритъмът за циклична нормализация на льос. Индексите и съответстват на числата на микрочипове, а индексът - на номера на проба или подмножество от проби, съответстващи на един ген.
Схема за извършване на циклична нормализация на льос
Литература
Gentleman R, Carey V, Huber W, Irizarry R, Dudoit S (ред.). Решения за биоинформатика и изчислителна биология, използващи R и Bioconductor. (2005) 473 p. ISBN: 978-0-387-25146-2.